News - Events
Latest news
A selection of recent relevant news
-
Published on
12th iGE3 Annual Meeting
Louis-Jeantet Foundation, Geneva - November 9, 2023 - Registration deadline: October 31, 2023

-
Published on
11th iGE3 PhD Awards - Call for application
For graduate students working in the laboratories of iGE3 members - Registration is closed

-
Published on
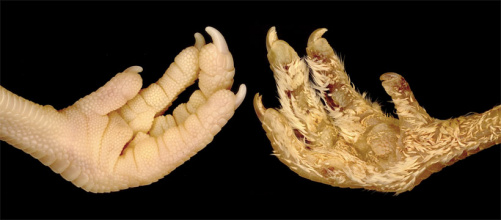
Scales or feathers? It all comes down to a few genes
Publication by Milinkovitch group
-
Published on
Gene responsible for severe facial defects identified
Publication by Antonarakis group
-
Published on
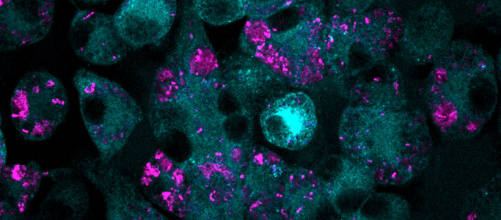
Bypassing antibiotic resistance with a combination of drugs
Publication by Kline group
-
Published on
Prix 3R 2023
Un des Prix 3R 2023 récompense Vladimir Katanaev

-
Published on
Prix Leenaards 2023
Un des trois Prix Leenaards 2023 décerné à Camilla Bellone

-
Published on
iGE3 conference - Silvia Arber
Neuronal circuits for body movements

-
Published on
New iGE3 member - Sophie Martin

-
Published on
New iGE3 member - Christoph Scheiermann

-
Published on
11th iGE3 Annual Meeting
Louis-Jeantet Foundation, Geneva - Hybrid via Zoom

-
Published on
10th iGE3 PhD Awards - Final selection

UNIGE Event Agenda
PerformanceCarouge et le Monde: les hautes écoles EXPLOREnt l'(extra)ordinaire
More information19.04.2024 – 20.04.2024 / Tours de Carouge et Marbrerie 13
ColloqueFemmes, écritures et expérience spirituelle : une question de genre ?
More information19.04.2024 09:30 – 17:00 / Auditorium Barbier-Müller, place du Bourg-de-Four 24
AnimationsStand de sensibilisation aux handicaps visuels, en collaboration avec la Fédération suisse des aveugles et malvoyants
More information19.04.2024 10:00 – 14:00 / Uni Mail / Hall d'Uni Mail
SéminaireBi-Factor and Second-Order Copula Models for Item Response Data
More information19.04.2024 11:15 – 12:15 / Uni Mail / Boulevard du Pont-d'Arve 40 1205 Geneva Room M 4220, 4th floor
SéminaireEarly- and late-time cosmology with Gravitational waves: latest results with a focus on topological defects
More information19.04.2024 11:50 – 12:50 / Ecole de Physique / EP234
Conférence - débatFlat Club - Christian Rüegg
More information19.04.2024 12:00 – 14:00 / Ecole de Physique / Auditoire Stueckelberg Pizza: 12:00, Start discussion: 12:30
SéminaireNeuroimmune Ecosystems
More information19.04.2024 12:15 – 13:00 / CMU / Jacques‐Louis Reverdin (B02.2526.a)
ColloqueGravitational waves in the nano-Hz band
More information19.04.2024 14:15 – 15:15 / Ecole de Physique / Auditoire Stuckelberg
Conférence - débatDécrypter les vagues de solidarités, XIXe - XXe siècles
More information19.04.2024 14:15 – 15:00 / ZOOM
ConférenceFestival histoire et cité: Dire le droit dans la rue
More information19.04.2024 14:30 – 16:00 / Uni Dufour / Auditoire U300
Soutenance de thèseOn the margins of procreative norms: life experiences and trajectories of childless people in India
More information19.04.2024 15:00 – 18:00 / Uni Mail / Salle M 6220
PerformanceBlue Marble Obsession
More information19.04.2024 18:30 – 20:00 / Chem. de la Marbrerie 13
ConcoursCONCOURS ROMAND D'ÉLOQUENCE 2024
More information19.04.2024 18:30 – 21:00 / Uni Dufour / Auditoire U600
Conférence - débatL’école face au péril écologique Entre responsabilité sociale et neutralité politique?
More information19.04.2024 19:00 – 21:00 / Uni Mail / Vendredi 19 avril 2024 19h00-20h45, Uni Mail salle MR150 Entrée libre Université de Genève, bâtiment d’Uni Mail 40 Bd du Pont-d’Arve, CH-1205 Genève
ExpositionPerspectives – L’univers mathématique de M.C. Escher
More information19.04.2024 07:30 – 19:00 / Bâtiment 66 bd Carl-Vogt / Salle d'exposition de l'UNIGE
AnimationsFestival Histoire et Cité 2024
More information19.04.2024 12:00 – 21:00 / Uni Dufour / divers
ExpositionÀ travers rues
More information15.04.2024 08:00 – 10.05.2024 17:00 / Uni Dufour, hall Rue du Général-Dufour 24, 1204 Genève
AnimationsHôpital des nounours 2024
More information15.04.2024 – 21.04.2024 / Salle communale du Faubourg, Rue des Terreaux-du-Temple 6, 1201 Genève










